一种微纤溶酶原变体及通过其得到的微纤溶酶变体的利记博彩app
本发明涉及在酶催化结构域具有一个或多个氨基酸突变的微纤溶酶原和微纤溶酶的变体,这些突变减少或防止了微纤溶酶的自身催化性蛋白水解。
背景技术:
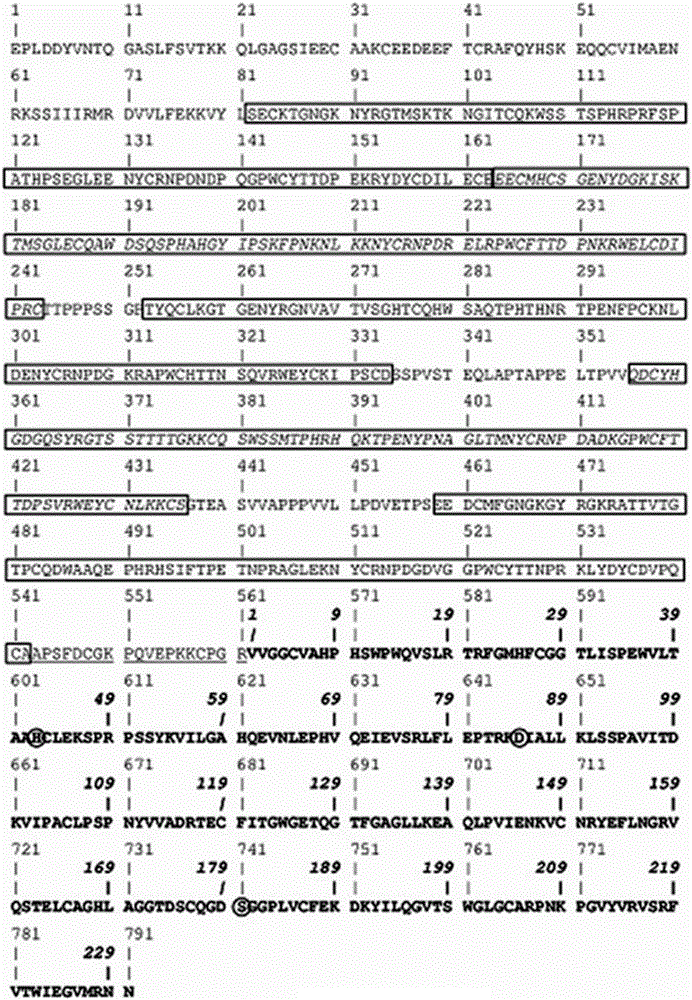
:纤溶蛋白溶解酶,简称纤溶酶(plasmin),是一种由纤溶酶原(plsminogen)激活而产生的丝氨酸蛋白酶。纤溶酶原是哺乳动物血液的重要组分,人纤溶酶原是由791个氨基酸组成的,分子量约为92KDa的单链糖蛋白。人纤溶酶原可以由纤溶酶原激活剂激活,纤溶酶原激活剂如尿激酶、链激酶、葡激酶、人组织型纤溶酶原激活剂(tPA)、或它们的变体。激活后,人纤溶酶原在Arg561-Val562处水解,形成由二硫键结合在一起的一条重链和一条轻链。重链(残基1-561,大约60kDa)包含5个Kringle区域(K1~K5),各自包含大约80个氨基酸残基;轻链(残基562-791,大约25kDa)包含催化结构域。Arg561-Val562处的水解诱导轻链发生大的构象变化,该变化导致轻链内的由His603,Asp646,Ser741构成的催化三联体的启动或激活。人纤溶酶原有多种截短形式,人小纤溶酶原包括人纤溶酶原的443~791位的氨基酸肽段,由第五个Kringle区域和丝氨酸蛋白酶结构域组成。小纤溶酶原能够以与纤溶酶原相同的方式被激活,形成小纤溶酶。人微小纤溶酶原(microplasminogen),简称人微纤溶酶原,是由人纤溶酶原的进一步截短形式,不包括纤溶酶原的N端氨基酸肽段(NTP)、K1~K5区,但保留了活性区,被激活后,形成人微小纤溶酶,具有纤溶酶的纤溶活性。纤溶酶可以作为一种直接的纤维蛋白溶解剂或血栓溶解剂,在机体内的凝血和纤溶反应中发挥重要作用,此外,纤溶酶也参与金属蛋白酶类和脂氧合酶类的激活作用,同时还与机体内细胞许多迁移相关的生理和病理过程相关,比如癌症、血管增生、卵子成熟、胚胎发生、伤口愈合以及病原侵入有关。此外,还有研究提示纤溶酶可作为玻璃体切除术的辅助物,或提取自体纤溶酶(APE)用作药理玻璃休切除术的药剂。综上所述,纤溶酶在机体内发挥十分重要的作用,能够用以治疗相关的疾病,具有很高的药用价值。但人纤溶酶原或人纤溶酶也有一些获取和应用障碍,首先APE的分离耗时耗力,而且成本昂贵,不易获取;其次人纤溶酶原分子量较大,利用一些宿主细胞如大肠杆菌、哺乳动物细胞等表达较困难,表达效率比较低;最后由于人纤溶酶的分子量较大,使其作为药物进行治疗机体疾病时扩散较慢,影响疗效。重组人纤溶酶原或人纤溶酶的截短形式能够从一定程度上解决以上所述困难。有研究报道使用人微纤溶酶用于治疗眼部疾病,或作为玻璃体切除术的辅助药物,在玻璃体的注射位置向玻璃体视网膜接触面的扩散不会受到太大阻碍(CN100577202C)。纤溶酶和多种蛋白酶一样,会发生自身催化性蛋白水解,即自溶降解。早在1960年代早期,确立了纤溶酶在酸性pH下较稳定。但是有报道称即使将纤溶酶保存在pH3.8条件下,仍有部分位点发生自催化降解,而pH再降低到一定程度,纤溶酶的自身催化降解作用会减弱,但会有酸水解因素导致其不稳定(W001/36608)。专利US5879923中提到另外一种方法是添加寡聚多肽复合物(oligopeptidiccompounds),可以稳定纤溶酶。专利WO93/15189提到将纤溶酶聚乙二醇化也可以增加纤溶酶稳定性。但是上述报道都没有从分子水平解决纤溶酶自溶降解的问题。有研究报道对纤溶酶原不同氨基酸位点进行突变,得到不同的效果,揭示了某些位点的相关作用。Dawson等人在专利WO91/08297中描述了不降解的纤溶酶原变体(重链和轻链之间的裂解位点被破坏)。Dawson等人用Glu取代719处的Arg(R719E)可以降低Glu-纤溶酶原变体与链激酶的亲和力(1994,Biochemistry33,12042-12047)。Jespers等人利用噬菌体展示技术筛选了微纤溶酶原单突变H569A,R610A,K615A,D660A,Y672A,R712A,R719A,T782A,R789A,发现719位点对于和链激酶的相互作用是关键的。Terzyan等人报道了在R561A突变的基础上进行了其他单突变(K698M,D740N,S741A),发现这些变异体能够阻止激活纤溶酶原的蛋白水解活性。从而进一步阻止活性的纤溶酶的形成(2004,Proteins56,277-284)。上述的研究报道的突变都没有降低纤溶酶的自溶降解程度。而降低纤溶酶的自溶降解程度,对于安全有效的长期保存,安全有效的治疗溶解血栓(thrombolytictherapy)或眼睛里的后玻璃体黏附(posteriorvitreousdetachment)或玻璃体液化(vitreousliquefaction)起到重要的作用。技术实现要素:本发明涉及人纤溶酶原序列的543-791位间249个氨基酸残基的微纤溶酶原,在酶催化结构域具有一个或多个氨基酸突变的微纤溶酶原变体,或由此而来的微纤溶酶变体,或独立的微纤溶酶变体。所述的变体其特征是在SEQIDNO:2序列的29位组氨酸(H)或/和103位的赖氨酸(K)突变为不易于自溶的氨基酸,在上述突变基础上,1-4位氨基酸可以一个或多个缺失或者不缺失。所述的微纤溶酶原变体的序列选自,但不限于以下表1中的序列表1微纤溶酶原野生型及变体序列本发明另一方面提供了由所述的微纤溶酶原变体得到的微纤溶酶变体或独立的与本发明所述的微纤溶酶原变体相对应的微纤溶酶变体。在本发明中,“微纤溶酶原变体得到的微纤溶酶变体”指的是经过纤溶酶原激活剂激活后将微纤溶酶原变体转化而成的微纤溶酶变体,独立的与本发明所述的微纤溶酶原变体相对应的微纤溶酶变体指的是不经过微纤溶酶原变体,而通过其它方式直接获得的与本发明所提供的微纤溶酶原变体相对应的微纤溶酶变体。所述的微纤溶酶原变体、微纤溶酶变体、或它们的衍生物,能降低自催化降解的程度。自催化降解程度是用产色底物或生物底物活性方法检测,并和野生型微纤溶酶的自催化降解的程度比较。进一步特征在于本发明的所述变体的自溶降解常数是野生型微纤溶酶的至多50%,并且它们的催化常数Kcat是在野生型纤溶酶的Kcat的50%至200%的范围内。本发明提供了纤溶酶原催化结构域特定位置的氨基酸突变,所述突变能够降低或防止纤溶酶自溶降解程度,所述突变不是仅限于本发明所述的微纤溶酶原或微纤溶酶,可以扩展为其他纤溶酶原或纤溶酶,可以是谷氨酰纤溶酶原(Glu-plasminogen)或谷氨酰纤溶酶(Glu-plasmin),赖氨酰纤溶酶原(Lys-plasminogen)或赖氨酰纤溶酶(Lys-plasmin),中纤溶酶原(midiplasminogen)或中纤溶酶(midiplasmin),小纤溶酶原(miniplasminogen)或小纤溶酶(miniplasmin),微纤溶酶原(microplasminogen)或微纤溶酶(microplasmin),delta纤溶酶原(deltaplasminogen)或delta纤溶酶(deltaplasmin)。本发明提供了获得所述微纤溶酶原变体的方法,所述方法包括以下步骤:(1)将编码所述微纤溶酶原变体的核酸连接到表达载体,导入能够表达所述微纤溶酶原的宿主细胞中,其中所述的宿主细胞优选为细菌,更优选为大肠杆菌,如BL21(DE3)、DH5α等。也优选为酵母菌,更优选为毕赤酵母,如GS115、X33等;也优选为酿酒酵母,如INVSc1等。(2)在合适的条件下使(1)中所述宿主菌表达微纤溶酶原;(3)收获(2)中表达的微纤溶酶原;(4)将(3)中的微纤溶酶原激活成微纤溶酶;本发明还涉及编码纤溶酶原变体或纤溶酶变体的核苷酸序列,含有这些核苷酸序列的重组载体。转化上述核苷酸序列或载体到宿主细胞也是本发明的一部分。所述的宿主细胞可选自细菌或酵母菌,所述细菌优选为大肠杆菌,所述酵母菌优选为毕赤酵母或酿酒酵母。本发明提供了微纤溶酶原变体、微纤溶酶变体、变体衍生物或任何组合形式的药物组合物,药物组合物包含常用的佐剂和添加剂,或药学上任何可接受的载体、稀释剂或稳定剂,且优选配制成溶液制剂。本发明提供了降低自溶降解程度的微纤溶酶原变体、微纤溶酶变体、变体衍生物或任何组合形式的药物组合物,用于诱导或促使受试着体内的病理性纤维蛋白沉积的溶解,或用于减少循环纤维蛋白原、或用于受试者的受损组织的酶清创术。本发明提供了降低自溶降解程度的微纤溶酶原变体、微纤溶酶变体、变体衍生物或任何形式的药物组合物,用于制备治疗眼部病症的药物中的用途,其中眼部病症选自如下疾病:视网膜脱离、视网膜破裂、玻璃体出血、糖尿病性玻璃体出血、增殖性糖尿病性视网膜病、非增殖性糖尿病性视网膜病、与衰老有关的黄斑变性、黄斑破洞、玻璃体黄斑牵引、黄斑皱褶、黄斑渗出物、囊状黄斑水肿、纤维蛋白沉积、视网膜静脉阻塞、视网膜动脉阻塞、视网膜下出血、弱视、眼内炎、早产儿视网膜病变、青光眼、视网膜色素变性以及以上任意的组合。本发明所用氨基酸三字母代码和单字母代码如J.biol.chem,243,p3558(1968)中所述。术语“SDS-PAGE电泳”是指十二烷基硫酸钠聚丙烯酰胺凝胶电泳,用于分离蛋白质。“HPLC”是指高效液相色谱法,是以液体为流动相,采用高压输液系统,将具有不同极性的单一溶剂或不同比例的混合溶剂、缓冲液等流动相泵入装有固定相的色谱柱,在柱内各成分被分离后,进入检测器进行检测,从而实现对试样的分析。“Kex2蛋白酶”是酵母自身编码的一种前体加工酶,是一个钙离子依赖型的丝氨酸蛋白酶,其专一性的切割Lys-Arg,Arg-Arg,Pro-Arg的C末端肽键。附图说明图1用于计数的野生型人Glu-纤溶酶(1-791)的氨基酸序列,以及纤溶酶的催化域(加粗显示的1-230位氨基酸),并标出了5个Kringle区域,本发明的野生型微纤溶酶原序列是543-791位氨基酸序列;图2重组微纤溶酶原的克隆方法示意图;图3微纤溶酶不同时长自溶降解SDS-PAGE电泳图,微纤溶酶原和微纤溶酶为27kDa左右,降解后会有15kDa左右和10kDa左右的杂质;图4A微纤溶酶原纯化后HPLC图谱;图4B微纤溶酶纯化后HPLC图谱。具体实施方式以下结合实施例用于进一步描述本发明,但这些实施例并非限制本发明的范围。实施例1:野生型微纤溶酶原及变体的构建和表达1.基因合成野生型人微纤溶酶原核酸序列由南京金斯瑞生物公司合成。合成的野生型微纤溶酶原核酸序列如SEQIDNO.1所示,在5’端加了一个EcoRI酶切位点和Kex2切割位点,并在3’端添加了一个NotI酶切位点。斜体部分分别是EcoRI和NotI酶切位点,下划线部分是Kex2切割位点,这个位点被Kex2识别并切割除去a-因子分泌信号肽,形成成熟的蛋白。对应的蛋白序列如下所示APSFDCGKPQVEPKKCPGRVVGGCVAHPHSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRKDIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNN2.野生型微纤溶酶原重组表达载体的构建本发明所使用的表达载体是购自Invitrogen公司的毕赤酵母分泌表达载体pPIC9K(Invitrogen,Cat.K1750-01)。将合成的野生型微纤溶酶原基因片段用T载体试剂盒(Takara,Cat.D103A)连接到T载体。用内切酶EcoRI和NotI对上述带有微纤溶酶原基因的T载体和pPIC9K表达载体进行双酶切消化,37℃酶切2h。采用快速胶回收试剂盒(WizardSVGelandPCRClean-UpSystem,Cat#A9281,Lot#0000066824,Progema)回收基因和载体片段,用T4连接酶进行连接,16℃连接过夜。连接反应体系如下:取10μl连接反应液与TOP10感受态细胞混匀,热激转化。涂布带有氨苄抗性的培养平板,挑取阳性克隆,抽提质粒并由南京金斯瑞公司测序验证,得到野生型微纤溶酶原MP的表达载体。3.在野生型微纤溶酶氨基酸序列中引入本发明中所述的氨基酸突变。在一种变体中,29位的组氨酸置换成天冬酰胺,将其对应的核苷酸序列79-81位处的密码子CAC变为AAC。突变后的核酸序列如下:GCTCCATCTTTTGATTGTGGTAAGCCACAAGTTGAACCAAAAAAGTGTCCAGGCAGAGTGGTTGGTGGTTGTGTTGCTAACCCACATTCTTGGCCTTGGCAAGTTTCTTTGCGTACTAGATTCGGTATGCATTTTTGTGGCGGTACTTTGATTTCTCCAGAATGGGTTTTGACTGCTGCTCATTGTTTGGAAAAGTCTCCAAGACCATCTTCTTACAAGGTTATTTTGGGTGCTCATCAGGAGGTCAACCTAGAACCTCATGTTCAAGAAATTGAAGTTTCCAGATTGTTTTTGGAACCAACTAGAAAGGACATCGCACTTCTTAAATTGTCTTCACCAGCTGTTATTACTGATAAAGTTATTCCAGCTTGTTTGCCATCTCCAAACTACGTTGTTGCTGATAGAACTGAATGTTTTATCACTGGTTGGGGTGAAACTCAAGGCACTTTTGGTGCCGGTTTGTTGAAGGAAGCTCAATTGCCTGTTATAGAAAACAAAGTTTGTAACAGATACGAATTTTTGAACGGTAGAGTTCAATCTACTGAATTGTGTGCTGGTCATTTGGCTGGTGGTACTGATTCTTGTCAAGGTGATAGTGGAGGTCCATTGGTCTGTTTTGAAAAAGATAAGTACATTTTGCAAGGTGTTACTTCTTGGGGTTTGGGTTGTGCTAGACCAAATAAACCAGGTGTTTACGTTAGAGTTTCTAGATTTGTCACTTGGATTGAGGGTGTTATGAGAAACAACTAASEQNOID.3其对应的氨基酸序列如下所示,即MP1变体序列:APSFDCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRKDIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.4在另一种变体中,将103位的赖氨酸置换成丙氨酸,将其对应的核苷酸序列314-316位处的密码子AAG变为GCT。突变后的核酸序列如下所示:GCTCCATCTTTTGATTGTGGTAAGCCACAAGTTGAACCAAAAAAGTGTCCAGGCAGAGTGGTTGGTGGTTGTGTTGCTCACCCACATTCTTGGCCTTGGCAAGTTTCTTTGCGTACTAGATTCGGTATGCATTTTTGTGGCGGTACTTTGATTTCTCCAGAATGGGTTTTGACTGCTGCTCATTGTTTGGAAAAGTCTCCAAGACCATCTTCTTACAAGGTTATTTTGGGTGCTCATCAGGAGGTCAACCTAGAACCTCATGTTCAAGAAATTGAAGTTTCCAGATTGTTTTTGGAACCAACTAGAAAGGACATCGCACTTCTTAAATTGTCTTCACCAGCTGTTATTACTGATAAAGTTATTCCAGCTTGTTTGCCATCTCCAAACTACGTTGTTGCTGATAGAACTGAATGTTTTATCACTGGTTGGGGTGAAACTCAAGGCACTTTTGGTGCCGGTTTGTTGAAGGAAGCTCAATTGCCTGTTATAGAAAACAAAGTTTGTAACAGATACGAATTTTTGAACGGTAGAGTTCAATCTACTGAATTGTGTGCTGGTCATTTGGCTGGTGGTACTGATTCTTGTCAAGGTGATAGTGGAGGTCCATTGGTCTGTTTTGAAAAAGATAAGTACATTTTGCAAGGTGTTACTTCTTGGGGTTTGGGTTGTGCTAGACCAAATAAACCAGGTGTTTACGTTAGAGTTTCTAGATTTGTCACTTGGATTGAGGGTGTTATGAGAAACAACTAASEQNOID.5其对应的氨基酸序列如下所示,即MP2变体序列:APSFDCGKPQVEPKKCPGRVVGGCVAHPHSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.6另外,将29位的组氨酸置换成天冬酰胺,103位的赖氨酸置换成丙氨酸;将其对应的核苷酸序列79-81位处的密码子CAC变为AAC;其对应的核苷酸序列314-316位处的密码子AAG变为GCT。突变后的核酸序列如下所示:GCTCCATCTTTTGATTGTGGTAAGCCACAAGTTGAACCAAAAAAGTGTCCAGGCAGAGTGGTTGGTGGTTGTGTTGCTAACCCACATTCTTGGCCTTGGCAAGTTTCTTTGCGTACTAGATTCGGTATGCATTTTTGTGGCGGTACTTTGATTTCTCCAGAATGGGTTTTGACTGCTGCTCATTGTTTGGAAAAGTCTCCAAGACCATCTTCTTACAAGGTTATTTTGGGTGCTCATCAGGAGGTCAACCTAGAACCTCATGTTCAAGAAATTGAAGTTTCCAGATTGTTTTTGGAACCAACTAGAAAGGACATCGCACTTCTTAAATTGTCTTCACCAGCTGTTATTACTGATAAAGTTATTCCAGCTTGTTTGCCATCTCCAAACTACGTTGTTGCTGATAGAACTGAATGTTTTATCACTGGTTGGGGTGAAACTCAAGGCACTTTTGGTGCCGGTTTGTTGAAGGAAGCTCAATTGCCTGTTATAGAAAACAAAGTTTGTAACAGATACGAATTTTTGAACGGTAGAGTTCAATCTACTGAATTGTGTGCTGGTCATTTGGCTGGTGGTACTGATTCTTGTCAAGGTGATAGTGGAGGTCCATTGGTCTGTTTTGAAAAAGATAAGTACATTTTGCAAGGTGTTACTTCTTGGGGTTTGGGTTGTGCTAGACCAAATAAACCAGGTGTTTACGTTAGAGTTTCTAGATTTGTCACTTGGATTGAGGGTGTTATGAGAAACAACTAASEQNOID.7其对应的氨基酸序列如下所示,即MP3变体序列:APSFDCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.8在上述突变体的基础上,将1位或前4位的氨基酸进行缺失,即29位的组氨酸突变为天冬酰胺或/和103位的赖氨酸突变为丙氨酸,同时缺失1位或前4位的氨基酸。在29位的组氨酸突变为天冬酰胺的同时缺失第1位丙氨酸,得到微纤溶酶原变体MP4,其氨基酸序列如下:PSFDCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRKDIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.9在29位的组氨酸突变为天冬酰胺的同时缺失前4位氨基酸,得到微纤溶酶原变体MP5,其氨基酸序列如下:DCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRKDIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.10在103位的赖氨酸突变为丙氨酸的同时缺失第1位丙氨酸,得到微纤溶酶原变体MP6,其氨基酸序列如下:PSFDCGKPQVEPKKCPGRVVGGCVAHPHSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.11在103位的赖氨酸突变为丙氨酸的同时缺失前4位氨基酸,得到微纤溶酶原变体MP7,其氨基酸序列如下:DCGKPQVEPKKCPGRVVGGCVAHPHSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQNOID.1229位H突变为N、103位K突变为A,并且缺失第1位丙氨酸,得到微纤溶酶原变体MP8,其氨基酸序列如下:PSFDCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQIDNO.1329位H突变为N、103位K突变为A,并且缺失前4位氨基酸,得到微纤溶酶原变体MP9,其氨基酸序列如下:DCGKPQVEPKKCPGRVVGGCVAHPNSWPWQVSLRTRFGMHFCGGTLISPEWVLTAAHCLEKSPRPSSYKVILGAHQEVNLEPHVQEIEVSRLFLEPTRADIALLKLSSPAVITDKVIPACLPSPNYVVADRTECFITGWGETQGTFGAGLLKEAQLPVIENKVCNRYEFLNGRVQSTELCAGHLAGGTDSCQGDSGGPLVCFEKDKYILQGVTSWGLGCARPNKPGVYVRVSRFVTWIEGVMRNNSEQIDNO.14在野生型微纤溶酶原表达载体基础上,用KODPCR法引入单突变或双突变来得到MP1-MP3微纤溶酶原变体的表达载体,KODPCR法是使用KOD试剂盒(TOYOBO,Cat.KOD-201)来进行的,引物如表2所示。表2微纤溶酶原不同位点突变所用引物序列以SEQIDNO.15和SEQIDNO.16为引物,以MP的表达载体为模板进行KODPCR,获得MP1表达载体,KODPCR体系如下:扩增程序:94℃2min,94℃30s,55℃30s,68℃10min,扩增25个循环68℃孵育10min结束PCR扩增程序PCR反应结束后,在PCR反应液(总量50μL)中加入2μL内切酶DpnI,混匀,37℃反应1h。取10μL反应液与TOP10感受态细胞混匀,热激转化,涂布Amp培养平板,挑取阳性克隆,抽提质粒,送南京金斯瑞公司进行测序验证,得到MP1的表达载体。以SEQIDNO.17和SEQIDNO.18为引物,以MP的表达载体为模板进行KODPCR,获得MP2表达载体,条件及方法与上述MP1表达载体获取方法一致。以SEQIDNO.15和SEQIDNO.16为引物,以MP2的表达载体为模板进行KODPCR,获得MP3表达载体,条件及方法与上述MP1表达载体获取方法一致。用PCR的方法引入野生型微纤溶酶原第一位或前4位氨基酸的缺失,得到MP4-MP9微纤溶酶原变体,引物如表3所示。表3微纤溶酶原不同位点氨基酸缺失所用引物序列以相应序列为引物,以MP的表达载体为模版进行PCR,PCR条件:94℃3min,94℃30s,59℃50s,72℃30s,扩增30个循环72℃,5min结束PCR扩增程序使用胶回收试剂盒回收PCR片段,用T载体试剂盒(Takara,Cat.D103A)连接到T载体。用内切酶EcoRI和NotI对上述带有微纤溶酶原变体基因的T载体和pPIC9K表达载体进行双酶切消化。采用快速胶回收试剂盒回收基因和载体片段,然后使用快速连接酶连接,转化到TOP10感受态细胞,涂布Amp抗性培养平板,挑取单菌落,抽提质粒测序分析。具体步骤如野生型微纤溶酶原表达载体的构建。4.野生型微纤溶酶原及变体的重组表达将上述重组表达载体转化到从invitrogen公司购买的GS115毕赤酵母菌株(invitrogen,Cat.K1750-01),构建得到微纤溶酶原变体的重组菌,将重组菌涂布在含G418抗性的YPD平板进行筛选。转化及筛选方法步骤参见invitrogen公司的毕赤酵母表达操作手册(invitrogen,Cat.K1750-01)。挑取G418筛选后的单菌落于50mLBMGY培养基,30℃,250rpm培养20-24h。检测OD600值,应处于2-6之间。用离心机(BeckmanCoulter)3000rpm离心5min收集细胞,然后用BMMY培养基重悬至OD600为1,培养基中无水甲醇(分析纯)终浓度为0.5%,30℃,250rpm培养72h,每12h补加1/200总体积的无水甲醇,每24h取样1mL,12000rpm离心5min,收集上清。SDS-PAGE(invitrogen,Cat.No.456-1083)电泳检测目的蛋白表达。实施例2:微纤溶酶原的5L发酵罐规模发酵5L规模发酵分如下4步进行。1.取2ml甘油菌接种至装有200mlYPG培养基(酵母粉10g/L,大豆蛋白胨20g/L,甘油20g/L)的1L摇瓶中,在30℃下250rpm振荡培养约22h,培养至菌种OD600为12左右。2.将培养好的种子液接种至5L发酵罐中进行发酵,使用2.5LBSM培养基(其组分为:85%磷酸26.7ml/L,CaSO4·2H2O1.2g/L,K2SO418.2g/L,MgSO47.3g/L,KOH4.1g/L,甘油40.0g/L,PTM14.4ml/L,25%氨水调pH5.8。PTM1:CuSO4·5H2O,6.0g/L;KI,0.08g/L;MnSO4·H2O,3.0g/L;H3BO3,0.02g/L;MoNa2O4·2H2O,0.2g/L;CoCl2,0.5g/L;ZnCl2,20.0g/L;FeSO4·7H2O,65.0g/L;生物素,0.2g/L;H2SO4,5.0ml/L),培养温度设定为30℃,通气量保持6L/min,调整搅拌转速使DO(溶解氧浓度)>20%,用25%氨水调节pH,维持pH在5.8。3.培养至甘油耗尽时,DO迅速升高,此时开始流加甘油补料培养基(50%甘油含1.2%PTM1),调整搅拌转速使DO(溶解氧浓度)>20%,用25%氨水调节pH,维持pH在5.8。培养至菌种OD600达到150左右时,停止甘油补料。4.甘油补料结束后,一次性添加无水甲醇46mL,使发酵液中甲醇浓度达到0.2%~0.3%。待菌种适应甲醇环境后,将培养温度设置为25℃,培养pH设置为5.8,开始甲醇诱导,补入甲醇补料培养基(无水甲醇含1.2%PTM1),根据溶氧反馈调整甲醇补料速率,维持DO(溶解氧浓度)>20%,诱导48h,发酵结束。实施例3:微纤溶酶原及微纤溶酶的纯化发酵培养液经过一步中空纤维澄清后又分别用一步阳离子交换层析和一步阴离子交换层析两个步骤的纯化程序获得微纤溶酶原。激活后的微纤溶酶经过疏水层析、浓缩、置换缓冲液三步得到纯化的微纤溶酶(CN100577202C)。实施例4:微纤溶酶原及其变体的激活将纤溶酶原稀释到0.5g/L-1.0g/L(18μM-36μM),纤溶酶原与葡激酶的摩尔比为(50-200):1,25℃反应30min,加入预冷硫酸铵与氨甲环酸,使硫酸铵终浓度为1M,氨甲环酸终浓度为0.1M,以便稳定微纤溶酶,存于冰上,及时纯化。通过测定微纤溶酶原初始浓度及激活后有活性的微纤溶酶的浓度,最终微纤溶酶原及变体激活率都在90%以上。实施例5:测定野生型微纤溶酶及变体的自溶降解常数自溶常数K是一个二级速率常数,采用如下的方法测定:1.使用重组葡激酶(ENZ-288,Prospec公司)激活纯化的微纤溶酶原变体,使之转化成活性的微纤溶酶。将微纤溶酶原稀释到0.2g/L-0.5g/L(7μM-18μM),纤溶酶原与葡激酶的摩尔比为50:1(质量之比为88:1),37℃反应10min,此时微纤溶酶原已完全激活成微纤溶酶,且发生极少的自溶降解反应。之后每隔一定时间(0min,5min,10min,20min,30min,1h,2h,3h,6h)取出100μL样品,加入200μl预冷5mM柠檬酸终止反应,置于冰上保存;2.用HPLC检测所有时间点样品的峰面积,计算剩余微纤溶酶的摩尔浓度(用野生型的微纤溶酶原纯化品建立浓度对应峰面积的标准曲线)。或将所取的微纤溶酶样品稀释后(微纤溶酶终浓度控制在10nM-30nM)加入96孔板中0.5mM显色底物Glu-Phe-Lys-pNA(S2403,Chromogenix,米兰)中,将96孔板放置于预热37℃的酶标仪中反应,选择405nm波长,每30s读取一次数据,读取10min。通过微纤溶酶对S2403的水解活性的下降程度,计算出剩余微纤溶酶的摩尔浓度。3.绘制剩余微纤溶酶的浓度(mol/L)随时间(s)的降解曲线,并利用非线性回归分析,将数据带入方程中,获得自溶降解常数k(M-1s-1)。公式1:在公式中,k是自溶降解常数(单位是M-1s-1),t是酶反应时间(单位是s),CMP是任何给定的时间点处微纤溶酶的浓度(单位是mol/L),CMP0是t=0处微纤溶酶的浓度。实验结果,本发明所述的微纤溶酶突变体的自溶降解常数k是野生型人微纤溶酶的10%-50%,说明本发明的微纤溶酶原及纤溶酶突变体可以降低纤溶酶的自催化降解程度。微纤溶酶及其变异体的自溶常数k值见表4。表4微纤溶酶及其变异体的自溶降解常数k值名称对应氨基酸序列号突变位置自溶降解常数k(M-1s-1)MPSEQIDNO:2野生型120MP1SEQIDNO:4H29N56MP2SEQIDNO:6K103A35MP3SEQIDNO:8H29NK103A11MP4SEQIDNO:9H29N,缺失1A42MP5SEQIDNO:10H29N,缺失1A2P3S4F39MP6SEQIDNO:11K103A,缺失1A37MP7SEQIDNO:12K103A,缺失1A2P3S4F30MP8SEQIDNO:13H29NK103A,缺失1A12MP9SEQIDNO:14H29NK103A,缺失1A2P3S4F10实施例6:测定微纤溶酶变体的蛋白水解活性原理:通过检测微纤溶酶对显色底物S-2403,的水解活性,来测定Km,Kcat。S-2403水解后,生成产物pNA(对硝基苯胺),导致在405nm处的吸光度增加,使用酶标仪(Bio-Tek)检测在405nm处吸光度的变化,再根据吸光度对应pNA的含量的标准曲线,计算初始反应速率,从而获得相关的酶动力学参数。为获得活性的微纤溶酶,使用葡激酶激活,葡激酶与纤溶酶原摩尔比为1:50,控制微纤溶酶原浓度在0.2g/L-0.5g/L(7μM-18μM),缓冲液体系为25mM磷酸氢二钠,50mMNaCl,pH7.0,37℃孵育10min,加入2倍体积的预冷的5mM柠檬酸,将pH降低到大约3,并保存于4℃,终止反应并使微纤溶酶稳定。设置底物S2403的不同浓度(2mM,1mM,0.5mM,0.25mM,0.125mM,0.0625mM,0.03125mM,0.015625mM),每个浓度的底物重复3孔。预热酶标仪到37℃,选择405nm波长,每30s读取一次数据,读取10min。每个浓度的S2403有3个复孔,吸光度取平均值,减去本底吸光值,根据吸光度对应pNA的含量的标准曲线,计算初始水解速率。再通过以下公式分析数据,获得微纤溶酶变体对显色底物S-2403的酶促反应动力学参数(Km,Kcat),其中CMP是通过HPLC检测的活性微纤溶酶的浓度(mol/L)公式2:公式3:在公式中,v是初始反应速率(单位是μmol/s),Km为米氏常数(单位是μM),Kcat是催化系数(单位是s-1),Vmax为酶促反应最大速率,CMP是任何给定的时间点处微纤溶酶的浓度,CMP0是t=0处微纤溶酶的浓度。结果表明,本发明所述的微纤溶酶变体的米氏常数Km是野生型微纤溶酶的96%-120%,说明本发明所述的微纤溶酶变体对底物结合能力与野生型相当。本发明所述的微纤溶酶变体的催化系数Kcat是野生型微纤溶酶的的70%-140%,说明本发明所述的微纤溶酶变体的催化效率跟野生型微纤溶酶没有明显的降低,甚至有提高。微纤溶酶及其变异体的酶促反应动力学参数(Km,Kcat)见下表表5微纤溶酶及其变异体的KmKcat值序列表<110>江苏恒瑞医药股份有限公司<120>一种微纤溶酶原变体及通过其得到的微纤溶酶变体<130>360268CG-350169<160>22<170>PatentInversion3.3<210>1<211>770<212>DNA<213>人工序列<400>1gaattcaagagagctccatcttttgattgtggtaagccacaagttgaaccaaaaaagtgt60ccaggcagagtggttggtggttgtgttgctcacccacattcttggccttggcaagtttct120ttgcgtactagattcggtatgcatttttgtggcggtactttgatttctccagaatgggtt180ttgactgctgctcattgtttggaaaagtctccaagaccatcttcttacaaggttattttg240ggtgctcatcaggaggtcaacctagaacctcatgttcaagaaattgaagtttccagattg300tttttggaaccaactagaaaggacatcgcacttcttaaattgtcttcaccagctgttatt360actgataaagttattccagcttgtttgccatctccaaactacgttgttgctgatagaact420gaatgttttatcactggttggggtgaaactcaaggcacttttggtgccggtttgttgaag480gaagctcaattgcctgttatagaaaacaaagtttgtaacagatacgaatttttgaacggt540agagttcaatctactgaattgtgtgctggtcatttggctggtggtactgattcttgtcaa600ggtgatagtggaggtccattggtctgttttgaaaaagataagtacattttgcaaggtgtt660acttcttggggtttgggttgtgctagaccaaataaaccaggtgtttacgttagagtttct720agatttgtcacttggattgagggtgttatgagaaacaactaagcggccgc770<210>2<211>249<212>PRT<213>人工序列<400>2AlaProSerPheAspCysGlyLysProGlnValGluProLysLysCys151015ProGlyArgValValGlyGlyCysValAlaHisProHisSerTrpPro202530TrpGlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGly354045ThrLeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGlu505560LysSerProArgProSerSerTyrLysValIleLeuGlyAlaHisGln65707580GluValAsnLeuGluProHisValGlnGluIleGluValSerArgLeu859095PheLeuGluProThrArgLysAspIleAlaLeuLeuLysLeuSerSer100105110ProAlaValIleThrAspLysValIleProAlaCysLeuProSerPro115120125AsnTyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGly130135140GluThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeu145150155160ProValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGly165170175ArgValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThr180185190AspSerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLys195200205AspLysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAla210215220ArgProAsnLysProGlyValTyrValArgValSerArgPheValThr225230235240TrpIleGluGlyValMetArgAsnAsn245<210>3<211>750<212>DNA<213>人工序列<400>3gctccatcttttgattgtggtaagccacaagttgaaccaaaaaagtgtccaggcagagtg60gttggtggttgtgttgctaacccacattcttggccttggcaagtttctttgcgtactaga120ttcggtatgcatttttgtggcggtactttgatttctccagaatgggttttgactgctgct180cattgtttggaaaagtctccaagaccatcttcttacaaggttattttgggtgctcatcag240gaggtcaacctagaacctcatgttcaagaaattgaagtttccagattgtttttggaacca300actagaaaggacatcgcacttcttaaattgtcttcaccagctgttattactgataaagtt360attccagcttgtttgccatctccaaactacgttgttgctgatagaactgaatgttttatc420actggttggggtgaaactcaaggcacttttggtgccggtttgttgaaggaagctcaattg480cctgttatagaaaacaaagtttgtaacagatacgaatttttgaacggtagagttcaatct540actgaattgtgtgctggtcatttggctggtggtactgattcttgtcaaggtgatagtgga600ggtccattggtctgttttgaaaaagataagtacattttgcaaggtgttacttcttggggt660ttgggttgtgctagaccaaataaaccaggtgtttacgttagagtttctagatttgtcact720tggattgagggtgttatgagaaacaactaa750<210>4<211>249<212>PRT<213>人工序列<400>4AlaProSerPheAspCysGlyLysProGlnValGluProLysLysCys151015ProGlyArgValValGlyGlyCysValAlaHisProAsnSerTrpPro202530TrpGlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGly354045ThrLeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGlu505560LysSerProArgProSerSerTyrLysValIleLeuGlyAlaHisGln65707580GluValAsnLeuGluProHisValGlnGluIleGluValSerArgLeu859095PheLeuGluProThrArgLysAspIleAlaLeuLeuLysLeuSerSer100105110ProAlaValIleThrAspLysValIleProAlaCysLeuProSerPro115120125AsnTyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGly130135140GluThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeu145150155160ProValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGly165170175ArgValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThr180185190AspSerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLys195200205AspLysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAla210215220ArgProAsnLysProGlyValTyrValArgValSerArgPheValThr225230235240TrpIleGluGlyValMetArgAsnAsn245<210>5<211>750<212>DNA<213>人工序列<400>5gctccatcttttgattgtggtaagccacaagttgaaccaaaaaagtgtccaggcagagtg60gttggtggttgtgttgctcacccacattcttggccttggcaagtttctttgcgtactaga120ttcggtatgcatttttgtggcggtactttgatttctccagaatgggttttgactgctgct180cattgtttggaaaagtctccaagaccatcttcttacaaggttattttgggtgctcatcag240gaggtcaacctagaacctcatgttcaagaaattgaagtttccagattgtttttggaacca300actagaaaggacatcgcacttcttaaattgtcttcaccagctgttattactgataaagtt360attccagcttgtttgccatctccaaactacgttgttgctgatagaactgaatgttttatc420actggttggggtgaaactcaaggcacttttggtgccggtttgttgaaggaagctcaattg480cctgttatagaaaacaaagtttgtaacagatacgaatttttgaacggtagagttcaatct540actgaattgtgtgctggtcatttggctggtggtactgattcttgtcaaggtgatagtgga600ggtccattggtctgttttgaaaaagataagtacattttgcaaggtgttacttcttggggt660ttgggttgtgctagaccaaataaaccaggtgtttacgttagagtttctagatttgtcact720tggattgagggtgttatgagaaacaactaa750<210>6<211>249<212>PRT<213>人工序列<400>6AlaProSerPheAspCysGlyLysProGlnValGluProLysLysCys151015ProGlyArgValValGlyGlyCysValAlaHisProHisSerTrpPro202530TrpGlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGly354045ThrLeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGlu505560LysSerProArgProSerSerTyrLysValIleLeuGlyAlaHisGln65707580GluValAsnLeuGluProHisValGlnGluIleGluValSerArgLeu859095PheLeuGluProThrArgAlaAspIleAlaLeuLeuLysLeuSerSer100105110ProAlaValIleThrAspLysValIleProAlaCysLeuProSerPro115120125AsnTyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGly130135140GluThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeu145150155160ProValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGly165170175ArgValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThr180185190AspSerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLys195200205AspLysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAla210215220ArgProAsnLysProGlyValTyrValArgValSerArgPheValThr225230235240TrpIleGluGlyValMetArgAsnAsn245<210>7<211>750<212>DNA<213>人工序列<400>7gctccatcttttgattgtggtaagccacaagttgaaccaaaaaagtgtccaggcagagtg60gttggtggttgtgttgctaacccacattcttggccttggcaagtttctttgcgtactaga120ttcggtatgcatttttgtggcggtactttgatttctccagaatgggttttgactgctgct180cattgtttggaaaagtctccaagaccatcttcttacaaggttattttgggtgctcatcag240gaggtcaacctagaacctcatgttcaagaaattgaagtttccagattgtttttggaacca300actagaaaggacatcgcacttcttaaattgtcttcaccagctgttattactgataaagtt360attccagcttgtttgccatctccaaactacgttgttgctgatagaactgaatgttttatc420actggttggggtgaaactcaaggcacttttggtgccggtttgttgaaggaagctcaattg480cctgttatagaaaacaaagtttgtaacagatacgaatttttgaacggtagagttcaatct540actgaattgtgtgctggtcatttggctggtggtactgattcttgtcaaggtgatagtgga600ggtccattggtctgttttgaaaaagataagtacattttgcaaggtgttacttcttggggt660ttgggttgtgctagaccaaataaaccaggtgtttacgttagagtttctagatttgtcact720tggattgagggtgttatgagaaacaactaa750<210>8<211>249<212>PRT<213>人工序列<400>8AlaProSerPheAspCysGlyLysProGlnValGluProLysLysCys151015ProGlyArgValValGlyGlyCysValAlaHisProAsnSerTrpPro202530TrpGlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGly354045ThrLeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGlu505560LysSerProArgProSerSerTyrLysValIleLeuGlyAlaHisGln65707580GluValAsnLeuGluProHisValGlnGluIleGluValSerArgLeu859095PheLeuGluProThrArgAlaAspIleAlaLeuLeuLysLeuSerSer100105110ProAlaValIleThrAspLysValIleProAlaCysLeuProSerPro115120125AsnTyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGly130135140GluThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeu145150155160ProValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGly165170175ArgValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThr180185190AspSerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLys195200205AspLysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAla210215220ArgProAsnLysProGlyValTyrValArgValSerArgPheValThr225230235240TrpIleGluGlyValMetArgAsnAsn245<210>9<211>248<212>PRT<213>人工序列<400>9ProSerPheAspCysGlyLysProGlnValGluProLysLysCysPro151015GlyArgValValGlyGlyCysValAlaHisProAsnSerTrpProTrp202530GlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGlyThr354045LeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGluLys505560SerProArgProSerSerTyrLysValIleLeuGlyAlaHisGlnGlu65707580ValAsnLeuGluProHisValGlnGluIleGluValSerArgLeuPhe859095LeuGluProThrArgLysAspIleAlaLeuLeuLysLeuSerSerPro100105110AlaValIleThrAspLysValIleProAlaCysLeuProSerProAsn115120125TyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGlyGlu130135140ThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuPro145150155160ValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGlyArg165170175ValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAsp180185190SerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLysAsp195200205LysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArg210215220ProAsnLysProGlyValTyrValArgValSerArgPheValThrTrp225230235240IleGluGlyValMetArgAsnAsn245<210>10<211>245<212>PRT<213>人工序列<400>10AspCysGlyLysProGlnValGluProLysLysCysProGlyArgVal151015ValGlyGlyCysValAlaHisProAsnSerTrpProTrpGlnValSer202530LeuArgThrArgPheGlyMetHisPheCysGlyGlyThrLeuIleSer354045ProGluTrpValLeuThrAlaAlaHisCysLeuGluLysSerProArg505560ProSerSerTyrLysValIleLeuGlyAlaHisGlnGluValAsnLeu65707580GluProHisValGlnGluIleGluValSerArgLeuPheLeuGluPro859095ThrArgLysAspIleAlaLeuLeuLysLeuSerSerProAlaValIle100105110ThrAspLysValIleProAlaCysLeuProSerProAsnTyrValVal115120125AlaAspArgThrGluCysPheIleThrGlyTrpGlyGluThrGlnGly130135140ThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuProValIleGlu145150155160AsnLysValCysAsnArgTyrGluPheLeuAsnGlyArgValGlnSer165170175ThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAspSerCysGln180185190GlyAspSerGlyGlyProLeuValCysPheGluLysAspLysTyrIle195200205LeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArgProAsnLys210215220ProGlyValTyrValArgValSerArgPheValThrTrpIleGluGly225230235240ValMetArgAsnAsn245<210>11<211>248<212>PRT<213>人工序列<400>11ProSerPheAspCysGlyLysProGlnValGluProLysLysCysPro151015GlyArgValValGlyGlyCysValAlaHisProHisSerTrpProTrp202530GlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGlyThr354045LeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGluLys505560SerProArgProSerSerTyrLysValIleLeuGlyAlaHisGlnGlu65707580ValAsnLeuGluProHisValGlnGluIleGluValSerArgLeuPhe859095LeuGluProThrArgAlaAspIleAlaLeuLeuLysLeuSerSerPro100105110AlaValIleThrAspLysValIleProAlaCysLeuProSerProAsn115120125TyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGlyGlu130135140ThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuPro145150155160ValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGlyArg165170175ValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAsp180185190SerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLysAsp195200205LysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArg210215220ProAsnLysProGlyValTyrValArgValSerArgPheValThrTrp225230235240IleGluGlyValMetArgAsnAsn245<210>12<211>245<212>PRT<213>人工序列<400>12AspCysGlyLysProGlnValGluProLysLysCysProGlyArgVal151015ValGlyGlyCysValAlaHisProHisSerTrpProTrpGlnValSer202530LeuArgThrArgPheGlyMetHisPheCysGlyGlyThrLeuIleSer354045ProGluTrpValLeuThrAlaAlaHisCysLeuGluLysSerProArg505560ProSerSerTyrLysValIleLeuGlyAlaHisGlnGluValAsnLeu65707580GluProHisValGlnGluIleGluValSerArgLeuPheLeuGluPro859095ThrArgAlaAspIleAlaLeuLeuLysLeuSerSerProAlaValIle100105110ThrAspLysValIleProAlaCysLeuProSerProAsnTyrValVal115120125AlaAspArgThrGluCysPheIleThrGlyTrpGlyGluThrGlnGly130135140ThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuProValIleGlu145150155160AsnLysValCysAsnArgTyrGluPheLeuAsnGlyArgValGlnSer165170175ThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAspSerCysGln180185190GlyAspSerGlyGlyProLeuValCysPheGluLysAspLysTyrIle195200205LeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArgProAsnLys210215220ProGlyValTyrValArgValSerArgPheValThrTrpIleGluGly225230235240ValMetArgAsnAsn245<210>13<211>248<212>PRT<213>人工序列<400>13ProSerPheAspCysGlyLysProGlnValGluProLysLysCysPro151015GlyArgValValGlyGlyCysValAlaHisProAsnSerTrpProTrp202530GlnValSerLeuArgThrArgPheGlyMetHisPheCysGlyGlyThr354045LeuIleSerProGluTrpValLeuThrAlaAlaHisCysLeuGluLys505560SerProArgProSerSerTyrLysValIleLeuGlyAlaHisGlnGlu65707580ValAsnLeuGluProHisValGlnGluIleGluValSerArgLeuPhe859095LeuGluProThrArgAlaAspIleAlaLeuLeuLysLeuSerSerPro100105110AlaValIleThrAspLysValIleProAlaCysLeuProSerProAsn115120125TyrValValAlaAspArgThrGluCysPheIleThrGlyTrpGlyGlu130135140ThrGlnGlyThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuPro145150155160ValIleGluAsnLysValCysAsnArgTyrGluPheLeuAsnGlyArg165170175ValGlnSerThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAsp180185190SerCysGlnGlyAspSerGlyGlyProLeuValCysPheGluLysAsp195200205LysTyrIleLeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArg210215220ProAsnLysProGlyValTyrValArgValSerArgPheValThrTrp225230235240IleGluGlyValMetArgAsnAsn245<210>14<211>245<212>PRT<213>人工序列<400>14AspCysGlyLysProGlnValGluProLysLysCysProGlyArgVal151015ValGlyGlyCysValAlaHisProAsnSerTrpProTrpGlnValSer202530LeuArgThrArgPheGlyMetHisPheCysGlyGlyThrLeuIleSer354045ProGluTrpValLeuThrAlaAlaHisCysLeuGluLysSerProArg505560ProSerSerTyrLysValIleLeuGlyAlaHisGlnGluValAsnLeu65707580GluProHisValGlnGluIleGluValSerArgLeuPheLeuGluPro859095ThrArgAlaAspIleAlaLeuLeuLysLeuSerSerProAlaValIle100105110ThrAspLysValIleProAlaCysLeuProSerProAsnTyrValVal115120125AlaAspArgThrGluCysPheIleThrGlyTrpGlyGluThrGlnGly130135140ThrPheGlyAlaGlyLeuLeuLysGluAlaGlnLeuProValIleGlu145150155160AsnLysValCysAsnArgTyrGluPheLeuAsnGlyArgValGlnSer165170175ThrGluLeuCysAlaGlyHisLeuAlaGlyGlyThrAspSerCysGln180185190GlyAspSerGlyGlyProLeuValCysPheGluLysAspLysTyrIle195200205LeuGlnGlyValThrSerTrpGlyLeuGlyCysAlaArgProAsnLys210215220ProGlyValTyrValArgValSerArgPheValThrTrpIleGluGly225230235240ValMetArgAsnAsn245<210>15<211>36<212>DNA<213>人工序列<400>15gagtggttggtggttgtgttgctaacccacattctt36<210>16<211>36<212>DNA<213>人工序列<400>16aagaatgtgggttagcaacacaaccaccaaccactc36<210>17<211>55<212>DNA<213>人工序列<400>17ccagattgtttttggaaccaactagagctgacatcgcacttcttaaattgtcttc55<210>18<211>55<212>DNA<213>人工序列<400>18gaagacaatttaagaagtgcgatgtcagctctagttggttccaaaaacaatctgg55<210>19<211>37<212>DNA<213>人工序列<400>19ccggaattcaagagaccatcttttgattgtggtaagc37<210>20<211>31<212>DNA<213>人工序列<400>20ggcaaatggcattctgacatcctattgatta31<210>21<211>34<212>DNA<213>人工序列<400>21ccggaattcaagagagattgtggtaagccacaag34<210>22<211>31<212>DNA<213>人工序列<400>22ggcaaatggcattctgacatcctattgatta31当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1